Редактирование РНК

Редакти́рование РНК (англ. RNA editing) — процесс, в ходе которого нуклеотиды в новосинтезированной РНК подвергаются химическим модификациям. Редактирование РНК также может включать вставку, делецию или замену нуклеотидов в молекуле РНК. Редактирование РНК — довольно редкий процесс, и типичные этапы процессинга мРНК (кэпирование, полиаденилирование, сплайсинг) обычно не рассматриваются как редактирование.
Редактированию подвергаются некоторые тРНК, рРНК, мРНК и микроРНК у эукариот и поражающих их вирусов, а также прокариот[1]. Редактирование РНК происходит в клеточном ядре и цитозоле, а также в митохондриях и пластидах. У позвоночных редактирование РНК происходит редко и обычно выражается во внесении лишь небольших изменений в молекулу РНК. У некоторых других организмов, напротив, редактирование РНК происходит так интенсивно, что в конечной мРНК остаётся лишь несколько неотредактированных нуклеотидов. Процессы редактирования РНК очень разнообразны и в ходе эволюции появились независимо. Редактирование РНК может приводить к превращению цитидина (C) в уридин (U) и аденозина (A) в инозин (I) в результате дезаминирования, а также присоединению к РНК новых нуклеотидов и удалению уже входящих в её состав. Редактирование РНК может так изменить мРНК, что поменяется аминокислотный состав кодируемого белка и он будет отличаться от полипептида, предсказанного по последовательности гена[2].
Редактирование путём вставки или делеции
[править | править код]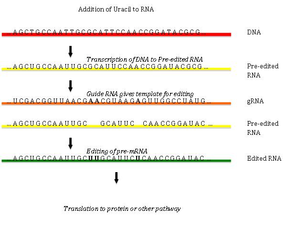
Редактирование РНК путём встраивания или удаления урацилов было описано в митохондриях кинетопластид Trypanosoma brucei[англ.][3][4][5][6][7][8][9][10]. Редактирование РНК начинается со спаривания первичного[англ.] нередактированного транскрипта с направляющей (гидовой) РНК, которая содержит комплементарные последовательности около сайтов встраивания или удаления. Образующийся двуцепочечный участок далее покрывается эдитосомой — крупным многобелковым комплексом, катализирующим редактирование РНК[4][5]. Эдитосома начинает встраивание уридинов по первому положению неспаренных нуклеотидов. Встраиваемые уридины образуют комплементарные связи с направляющей РНК, и встраивание продолжается, пока в направляющей РНК встречаются A или G, и останавливается при появлении C или U[6][7]. Встраиваемые нуклеотиды вызывают сдвиг рамки считывания и приводят к тому, что транслируемый белок отличается от кодирующей области гена.
В ходе редактирования происходит разрезание по сайту, где не образуются комплементарные пары между направляющей РНК и нередактированным транскриптом. Следующая стадия катализируется ферментом концевой уридинтрансферазой, которая добавляет U из UTP к 3'-концу мРНК[8]. «Открытые» концы удерживаются другими белками эдитосомного комплекса. Другой фермент, U-специфичная экзорибонуклеаза, удаляет неспаренные уридины. После того как эдитосомный комплекс делает последовательность мРНК комплементарной направляющей РНК, РНК-лигаза соединяет концы отредактированной мРНК[9]. Эдитосомный комплекс способен редактировать мРНК лишь в направлении от 3'-конца к 5'-концу. Комплекс способен редактировать только по одной РНК в момент времени. РНК, для которой требуется значительное редактирование, нуждается в нескольких направляющих РНК и нескольких эдитосомных комплексах.
Редактирование посредством дезаминирования
[править | править код]Редактирование C → U
[править | править код]
Под действием фермента цитидиндезаминазы[англ.] происходит реакция дезаминирования, превращающая цитидин в урацил. Редактирование C → U может быть рассмотрено на примере гена аполипопротеина B у человека. В печени экспрессируется его изоформа Apo B100, а в кишечнике — apo B48. В клетках кишечника мРНК аполипопротеина B подвергается C → U редактированию, из-за которого кодон CAA превращается в стоп-кодон UAA, и синтезируется изоформа apo B48. Редактирование C → U часто происходит в митохондриальных РНК цветковых растений. У разных растений интенсивность редактирования C → U варьирует: у мха Funaria hygrometrica[англ.] в митохондриальных РНК происходит восемь актов редактирования, а у плауна Isoetes engelmannii[англ.] — около 1700[11]. Превращение C → U осуществляется семейством белков с пентатрикопептидными повторами (англ. pentatricopeptide repeat, PPR). Это семейство богато представлено у цветковых растений: так, у Arabidopsis насчитывается 450 белков этого семейства. Белки PPR были описаны также в пластидах и митохондриях[12].
Редактирование A → I
[править | править код]На долю превращений аденозина в инозин (A → I) приходится около 90 % всех случаев редактирования РНК. Дезаминирование аденозина катализируется аденозиндезаминазой, специфичной к двуцепочечной РНК[англ.] (англ. double-stranded RNA-specific adenosine deaminase, ADAR), которые обычно действуют на предшественников мРНК (пре-мРНК). Дезаминирование аденозина с образованием инозина разрушает спаривание оснований в двуцепочечной РНК (дцРНК), поэтому некоторые двуцепочечные РНК дают начало меньшему количеству малых интерферирующих РНК, чем другие. В дезаминированной дцРНК формируются wobble-взаимодействия между парами оснований, из-за чего молекула приобретает необычную структуру, подавляющую инициацию трансляции. РНК, содержащая пары U — I, привлекает метилазы[англ.], участвующие в образовании гетерохроматина, кроме того, сайты редактирования A → I часто совпадают с сайтами связывания с микроРНК, что создаёт конкуренцию между двумя процессами[13]. Редактирование A → I активно изучается в связи с концепцией эпитранскриптомики[англ.], которая гласит, что химические модификации РНК могут влиять на её функции[14][15]. Когда в ходе трансляции рибосома встречается с инозином, она распознаёт его как гуанин, хотя некоторые исследования свидетельствуют, что I может считываться как A и U. Кроме того, показано, что рибосома замедляется, когда встречает инозин в мРНК[16].
Интенсивность редактирования A → I может быть тканеспецифичной, как в случае филамина A[англ.] человека[17]. В числе факторов, влияющих на интенсивность редактирования, называют эффективность сплайсинга пре-мРНК[18].
В связи с интенсивным развитием технологий высокопроизводительного секвенирования стало возможным создание баз данных, содержащих сведения о редактировании различных РНК. В 2013 году был открыт каталог RADAR (от англ. Rigorously Annotated Database of A-to-I RNA editing), содержащий сайты редактирования A → I, а также данные о таких тканеспецифичных заменах у человека, мыши и дрозофилы. В базу постоянно заносятся новооткрытые сайты редактирования[19].
Альтернативное редактирование мРНК
[править | править код]В случае гена WT1[англ.] было описано альтернативное редактирование РНК U → C[20], кроме того, случаи неканонического редактирования G → A известны для транскриптов гетерогенного ядерного рибонуклеопротеина[англ.] K в нормальных и злокачественных клетках толстой кишки[21]. Редактирование G → A было также отмечено, наряду с неклассическим превращением U → C, в транскриптах триптофангидроксилазы 2 в нейронах[22]. Хотя наиболее простым механизмом превращения U → C может быть обратное аминирование, предполагается, что в митохондриальных транскриптах в основе редактирования U → C лежат реакции трансаминирования и трансгликозилирования[23]. В середине 2010-х годов исследование редактирования G → A в транскрипте WT1 показало, что это превращение наиболее активно происходит в двух точках под действием фермента APOBEC3A[англ.] (каталитический полипептид 3A фермента редактирования мРНК аполипопротеина B)[24].
Редактирование РНК в митохондриях и пластидах растений
[править | править код]Многочисленные исследования показали, что в митохондриях растений РНК подвергается только редактированию C → U и, очень редко, U → C[25][26][27][28][29][30][31][32][33][34][35][36][37]. Сайты редактирования находятся преимущественно в кодирующих областях мРНК, интронах и других нетранслируемых областях[27]. Редактирование РНК может быть необходимым для восстановления функциональности молекул тРНК[29][30]. Хотя сайты редактирования C → U в пластидах и митохондриях изучены относительно хорошо[38], все белки, образующие соответствующие эдитосомы, ещё не идентифицированы. Показано, что в распознавании сайтов редактирования участвуют члены многочисленного белкового семейства PPR[39]. Для редактирования в некоторых сайтах необходимы белки семейства MORF (от англ. Multiple Organellar RNA editing Factor). Так как некоторые белки семейства MORF взаимодействуют с членами семейства PPR, возможно, что белки MORF входят в состав эдитосомы[40]. Ферменты, ответственные за дезаминирование и трансаминирование в органеллах, ещё не определены, но, возможно, эти реакции осуществляют члены семейства PPR. Редактирование РНК необходимо для нормального клеточного дыхания и трансляции в клетках растений. Редактирование антикодоновой петли может восстанавливать функциональность молекул тРНК[41].
Редактирование РНК у вирусов
[править | править код]Некоторые вирусы, такие как возбудители кори, свинки[англ.] и парагриппа, используют редактирование РНК для получения новых вариантов белков[42][43]. Вирусные РНК синтезируют кодируемые вирусами РНК-зависимые РНК-полимеразы, которые иногда «запинаются» на определённых сочетаниях нуклеотидов. Остановка РНК-полимеразы может привести к вставке дополнительных гуаниновых или адениноных нуклеотидов. Вставка дополнительных нуклеотидов смещает рамку считывания, что приводит к образованию новых форм белков. Кроме того, к 3'-концу созревающих вирусных мРНК может добавляться до нескольких сотен дополнительных адениновых нуклеотидов, которые стабилизируют мРНК[44].
Функции
[править | править код]Редактирование РНК может выполнять несколько функций. В частности, оно может быть связано с деградацией РНК. В 2008 году было показано, что ADAR и UPF1[англ.] (фермент, участвующий в нонсенс-опосредованном распаде) взаимодействуют друг с другом и со сплайсосомой, формируя супрасплайсосому, и могут подавлять экспрессию некоторых генов. Подобно альтернативному сплайсингу, редактирование РНК может приводить к появлению новых форм белков за счёт замен, а также появления или удаления сайтов сплайсинга. Редактирование некодирующих РНК может изменить их структуру или привести к новым мутациям в вирусных геномах. Редактирование РНК может также представлять собой механизм защиты от ретротранспозонов[45].

Происхождение и эволюция
[править | править код]У истоков редактирования РНК животных могли стоять мононуклеотиддезаминазы, которые дали начало обширным белковым семействам, включающим такие ферменты редактирования РНК, как ADAR и APOBEC1[англ.]. Последовательности этих генов сближают их с бактериальными дезаминазами, участвующими в метаболизме нуклеотидов. Аденозиндезаминаза кишечной палочки Escherichia coli не может катализировать дезаминирование нуклеозида в РНК: её «карман», в котором и происходит реакция, слишком мал, чтобы вместить целую молекулу РНК. Однако расширение активного сайта наблюдается в человеческих белках APOBEC1 и ADAR, которые могут катализировать дезаминирование РНК[46][47]. Масштабное редактирование РНК, зависимое от направляющих РНК, такое как вставка нескольких уридиновых нуклеотидов у трипаносом, представляет собой совершенно иную биохимическую реакцию. Ферменты, ответственные за этот процесс, произошли от абсолютно других предшественников[4][48]. Однако специфичность вставки нуклеотидов, определяемая взаимодействием направляющей РНК с мРНК, схожа с процессом редактирования тРНК, протекающим в митохондриях животных и амёб Acanthamoeba[англ.][49]. Более того, этот процесс схож с метилированием рибозы в рРНК с участием направляющих РНК, которое происходит у всех эукариот[50].
Кардинально различающиеся пути редактирования РНК свидетельствуют, что они возникали независимо в ходе эволюции[51]. В некоторых источниках редактирование РНК рассматривают как процесс, направленный на устранение дефектов в последовательностях генов или их компенсацию[52].
См. также
[править | править код]Примечания
[править | править код]- ↑ Su A. A., Randau L. A-to-I and C-to-U editing within transfer RNAs. (англ.) // Biochemistry. Biokhimiia. — 2011. — August (vol. 76, no. 8). — P. 932—937. — doi:10.1134/S0006297911080098. — PMID 22022967.
- ↑ Brennicke A., Marchfelder A., Binder S. RNA editing. (англ.) // FEMS Microbiology Reviews. — 1999. — June (vol. 23, no. 3). — P. 297—316. — doi:10.1111/j.1574-6976.1999.tb00401.x. — PMID 10371035.
- ↑ Benne R. RNA editing in trypanosomes. (англ.) // European Journal Of Biochemistry. — 1994. — 1 April (vol. 221, no. 1). — P. 9—23. — doi:10.1111/j.1432-1033.1994.tb18710.x. — PMID 7513284.
- ↑ 1 2 3 Arts G. J., Benne R. Mechanism and evolution of RNA editing in kinetoplastida. (англ.) // Biochimica Et Biophysica Acta. — 1996. — 3 June (vol. 1307, no. 1). — P. 39—54. — doi:10.1016/0167-4781(96)00021-8. — PMID 8652667.
- ↑ 1 2 Alfonzo J. D., Thiemann O., Simpson L. The mechanism of U insertion/deletion RNA editing in kinetoplastid mitochondria. (англ.) // Nucleic Acids Research. — 1997. — 1 October (vol. 25, no. 19). — P. 3751—3759. — doi:10.1093/nar/25.19.3751. — PMID 9380494.
- ↑ 1 2 Blum B., Bakalara N., Simpson L. A model for RNA editing in kinetoplastid mitochondria: "guide" RNA molecules transcribed from maxicircle DNA provide the edited information. (англ.) // Cell. — 1990. — 26 January (vol. 60, no. 2). — P. 189—198. — doi:10.1016/0092-8674(90)90735-w. — PMID 1688737.
- ↑ 1 2 Kable M. L., Heidmann S., Stuart K. D. RNA editing: getting U into RNA. (англ.) // Trends In Biochemical Sciences. — 1997. — May (vol. 22, no. 5). — P. 162—166. — PMID 9175474.
- ↑ 1 2 Simpson L., Thiemann O. H. Sense from nonsense: RNA editing in mitochondria of kinetoplastid protozoa and slime molds. (англ.) // Cell. — 1995. — 16 June (vol. 81, no. 6). — P. 837—840. — doi:10.1016/0092-8674(95)90003-9. — PMID 7781060.
- ↑ 1 2 Stuart K. RNA editing in mitochondrial mRNA of trypanosomatids. (англ.) // Trends In Biochemical Sciences. — 1991. — February (vol. 16, no. 2). — P. 68—72. — PMID 1713359.
- ↑ van der Spek H., Arts G. J., Zwaal R. R., van den Burg J., Sloof P., Benne R. Conserved genes encode guide RNAs in mitochondria of Crithidia fasciculata. (англ.) // The EMBO Journal. — 1991. — May (vol. 10, no. 5). — P. 1217—1224. — PMID 1708723.
- ↑ Takenaka M., Verbitskiy D., Zehrmann A., Härtel B., Bayer-Császár E., Glass F., Brennicke A. RNA editing in plant mitochondria—connecting RNA target sequences and acting proteins. (англ.) // Mitochondrion. — 2014. — November (vol. 19 Pt B). — P. 191—197. — doi:10.1016/j.mito.2014.04.005. — PMID 24732437.
- ↑ Shikanai T. RNA editing in plants: Machinery and flexibility of site recognition. (англ.) // Biochimica Et Biophysica Acta. — 2015. — September (vol. 1847, no. 9). — P. 779—785. — doi:10.1016/j.bbabio.2014.12.010. — PMID 25585161.
- ↑ Nishikura K. Functions and regulation of RNA editing by ADAR deaminases. (англ.) // Annual Review Of Biochemistry. — 2010. — Vol. 79. — P. 321—349. — doi:10.1146/annurev-biochem-060208-105251. — PMID 20192758.
- ↑ Tajaddod M., Jantsch M. F., Licht K. The dynamic epitranscriptome: A to I editing modulates genetic information. (англ.) // Chromosoma. — 2016. — March (vol. 125, no. 1). — P. 51—63. — doi:10.1007/s00412-015-0526-9. — PMID 26148686.
- ↑ Licht K., Jantsch M. F. Rapid and dynamic transcriptome regulation by RNA editing and RNA modifications. (англ.) // The Journal Of Cell Biology. — 2016. — 11 April (vol. 213, no. 1). — P. 15—22. — doi:10.1083/jcb.201511041. — PMID 27044895.
- ↑ Licht K., Hartl M., Amman F., Anrather D., Janisiw M. P., Jantsch M. F. Inosine induces context-dependent recoding and translational stalling. (англ.) // Nucleic Acids Research. — 2019. — 10 January (vol. 47, no. 1). — P. 3—14. — doi:10.1093/nar/gky1163. — PMID 30462291.
- ↑ Stulić M., Jantsch M. F. Spatio-temporal profiling of Filamin A RNA-editing reveals ADAR preferences and high editing levels outside neuronal tissues. (англ.) // RNA Biology. — 2013. — October (vol. 10, no. 10). — P. 1611—1617. — doi:10.4161/rna.26216. — PMID 24025532.
- ↑ Licht K., Kapoor U., Mayrhofer E., Jantsch M. F. Adenosine to Inosine editing frequency controlled by splicing efficiency. (англ.) // Nucleic Acids Research. — 2016. — 27 July (vol. 44, no. 13). — P. 6398—6408. — doi:10.1093/nar/gkw325. — PMID 27112566.
- ↑ Ramaswami G., Li J. B. RADAR: a rigorously annotated database of A-to-I RNA editing. (англ.) // Nucleic Acids Research. — 2014. — January (vol. 42). — P. D109—113. — doi:10.1093/nar/gkt996. — PMID 24163250.
- ↑ Sharma P. M., Bowman M., Madden S. L., Rauscher FJ 3rd., Sukumar S. RNA editing in the Wilms' tumor susceptibility gene, WT1. (англ.) // Genes & Development. — 1994. — 15 March (vol. 8, no. 6). — P. 720—731. — doi:10.1101/gad.8.6.720. — PMID 7926762.
- ↑ Klimek-Tomczak K., Mikula M., Dzwonek A., Paziewska A., Karczmarski J., Hennig E., Bujnicki J. M., Bragoszewski P., Denisenko O., Bomsztyk K., Ostrowski J. Editing of hnRNP K protein mRNA in colorectal adenocarcinoma and surrounding mucosa. (англ.) // British Journal Of Cancer. — 2006. — 27 February (vol. 94, no. 4). — P. 586—592. — doi:10.1038/sj.bjc.6602938. — PMID 16404425.
- ↑ Grohmann M., Hammer P., Walther M., Paulmann N., Büttner A., Eisenmenger W., Baghai T. C., Schüle C., Rupprecht R., Bader M., Bondy B., Zill P., Priller J., Walther D. J. Alternative splicing and extensive RNA editing of human TPH2 transcripts. (англ.) // PloS One. — 2010. — 29 January (vol. 5, no. 1). — P. e8956—8956. — doi:10.1371/journal.pone.0008956. — PMID 20126463.
- ↑ Castandet B., Araya A. RNA editing in plant organelles. Why make it easy? (англ.) // Biochemistry. Biokhimiia. — 2011. — August (vol. 76, no. 8). — P. 924—931. — doi:10.1134/S0006297911080086. — PMID 22022966.
- ↑ Niavarani A., Currie E., Reyal Y., Anjos-Afonso F., Horswell S., Griessinger E., Luis Sardina J., Bonnet D. APOBEC3A is implicated in a novel class of G-to-A mRNA editing in WT1 transcripts. (англ.) // PloS One. — 2015. — Vol. 10, no. 3. — P. e0120089—0120089. — doi:10.1371/journal.pone.0120089. — PMID 25807502.
- ↑ Covello P. S., Gray M. W. RNA editing in plant mitochondria. (англ.) // Nature. — 1989. — 19 October (vol. 341, no. 6243). — P. 662—666. — doi:10.1038/341662a0. — PMID 2552326.
- ↑ Gualberto J. M., Lamattina L., Bonnard G., Weil J. H., Grienenberger J. M. RNA editing in wheat mitochondria results in the conservation of protein sequences. (англ.) // Nature. — 1989. — 19 October (vol. 341, no. 6243). — P. 660—662. — doi:10.1038/341660a0. — PMID 2552325.
- ↑ 1 2 Hiesel R., Wissinger B., Schuster W., Brennicke A. RNA editing in plant mitochondria. (англ.) // Science (New York, N.Y.). — 1989. — 22 December (vol. 246, no. 4937). — P. 1632—1634. — doi:10.1126/science.2480644. — PMID 2480644.
- ↑ Hoch B., Maier R. M., Appel K., Igloi G. L., Kössel H. Editing of a chloroplast mRNA by creation of an initiation codon. (англ.) // Nature. — 1991. — 12 September (vol. 353, no. 6340). — P. 178—180. — doi:10.1038/353178a0. — PMID 1653905.
- ↑ 1 2 Pring D., Brennicke A., Schuster W. RNA editing gives a new meaning to the genetic information in mitochondria and chloroplasts. (англ.) // Plant Molecular Biology. — 1993. — March (vol. 21, no. 6). — P. 1163—1170. — PMID 8490134.
- ↑ 1 2 Wissinger B., Brennicke A., Schuster W. Regenerating good sense: RNA editing and trans splicing in plant mitochondria. (англ.) // Trends In Genetics : TIG. — 1992. — September (vol. 8, no. 9). — P. 322—328. — PMID 1365399.
- ↑ Yan J., Zhang Q., Yin P. RNA editing machinery in plant organelles. (англ.) // Science China. Life Sciences. — 2018. — February (vol. 61, no. 2). — P. 162—169. — doi:10.1007/s11427-017-9170-3. — PMID 29075943.
- ↑ Malek O., Lättig K., Hiesel R., Brennicke A., Knoop V. RNA editing in bryophytes and a molecular phylogeny of land plants. (англ.) // The EMBO Journal. — 1996. — 15 March (vol. 15, no. 6). — P. 1403—1411. — PMID 8635473.
- ↑ Freyer R., Kiefer-Meyer M. C., Kössel H. Occurrence of plastid RNA editing in all major lineages of land plants. (англ.) // Proceedings Of The National Academy Of Sciences Of The United States Of America. — 1997. — 10 June (vol. 94, no. 12). — P. 6285—6290. — doi:10.1073/pnas.94.12.6285. — PMID 9177209.
- ↑ Dietrich A., Small I., Cosset A., Weil J. H., Maréchal-Drouard L. Editing and import: strategies for providing plant mitochondria with a complete set of functional transfer RNAs. (англ.) // Biochimie. — 1996. — Vol. 78, no. 6. — P. 518—529. — PMID 8915541.
- ↑ Bock R., Hermann M., Fuchs M. Identification of critical nucleotide positions for plastid RNA editing site recognition. (англ.) // RNA (New York, N.Y.). — 1997. — October (vol. 3, no. 10). — P. 1194—1200. — PMID 9326494.
- ↑ Gray M. W., Covello P. S. RNA editing in plant mitochondria and chloroplasts. (англ.) // FASEB Journal : Official Publication Of The Federation Of American Societies For Experimental Biology. — 1993. — January (vol. 7, no. 1). — P. 64—71. — doi:10.1096/fasebj.7.1.8422976. — PMID 8422976.
- ↑ Marchfelder A., Binder S., Brennicke A., Knoop V. Preface // Modification and Editing of RNA (неопр.) / Grosjean H., Benne R.. — Washington, DC: ASM Press[англ.], 1998. — С. 307—323.
- ↑ Takenaka M., Zehrmann A., Verbitskiy D., Härtel B., Brennicke A. RNA editing in plants and its evolution. (англ.) // Annual Review Of Genetics. — 2013. — Vol. 47. — P. 335—352. — doi:10.1146/annurev-genet-111212-133519. — PMID 24274753.
- ↑ Barkan A., Small I. Pentatricopeptide repeat proteins in plants. (англ.) // Annual Review Of Plant Biology. — 2014. — Vol. 65. — P. 415—442. — doi:10.1146/annurev-arplant-050213-040159. — PMID 24471833.
- ↑ Bentolila S., Oh J., Hanson M. R., Bukowski R. Comprehensive high-resolution analysis of the role of an Arabidopsis gene family in RNA editing. (англ.) // PLoS Genetics. — 2013. — June (vol. 9, no. 6). — P. e1003584—1003584. — doi:10.1371/journal.pgen.1003584. — PMID 23818871.
- ↑ Price D. H., Gray M. W. Editing of tRNA // Modification and Editing of RNA (неопр.) / Grosjean H., Benne R.. — Washington, DC: ASM Press[англ.], 1998. — С. 289—306.
- ↑ Curran J., Boeck R., Kolakofsky D. The Sendai virus P gene expresses both an essential protein and an inhibitor of RNA synthesis by shuffling modules via mRNA editing. (англ.) // The EMBO Journal. — 1991. — October (vol. 10, no. 10). — P. 3079—3085. — PMID 1655410.
- ↑ Zheng H., Fu T. B., Lazinski D., Taylor J. Editing on the genomic RNA of human hepatitis delta virus. (англ.) // Journal Of Virology. — 1992. — August (vol. 66, no. 8). — P. 4693—4697. — PMID 1629949.
- ↑ Kolakofsky D., Hausmann S. Chapter 23: Cotranscriptional Paramyxovirus mRNA Editing: a Contradiction in Terms? // Modification and Editing of RNA (неопр.) / Grosjean H., Benne R.. — Washington, DC: ASM Press[англ.], 1998. — С. 413—420.
- ↑ RNA editing by John W. Kimball. Дата обращения: 11 августа 2019. Архивировано 11 августа 2019 года.
- ↑ Carter C. W. Nucleoside deaminases for cytidine and adenosine: comparisons with deaminases acting on RNA // Modification and Editing of RNA (неопр.) / Grosjean H., Benne R.. — Washington, DC: ASM Press[англ.], 1998. — С. 363—376.
- ↑ Covello P. S., Gray M. W. On the evolution of RNA editing. (англ.) // Trends In Genetics : TIG. — 1993. — August (vol. 9, no. 8). — P. 265—268. — PMID 8379005.
- ↑ Lonergan K. M., Gray M. W. Predicted editing of additional transfer RNAs in Acanthamoeba castellanii mitochondria. (англ.) // Nucleic Acids Research. — 1993. — 11 September (vol. 21, no. 18). — P. 4402—4402. — doi:10.1093/nar/21.18.4402. — PMID 8415006.
- ↑ Bachellerie J. P., Cavaille J. Small nucleolar RNAs guide the ribose methylations of eukaryotic rRNAs // Modification and Editing of RNA (неопр.) / Grosjean H., Benne R.. — Washington, DC: ASM Press[англ.], 1998. — С. 255—272.
- ↑ Speijer D. Does constructive neutral evolution play an important role in the origin of cellular complexity? Making sense of the origins and uses of biological complexity. (англ.) // BioEssays : News And Reviews In Molecular, Cellular And Developmental Biology. — 2011. — May (vol. 33, no. 5). — P. 344—349. — doi:10.1002/bies.201100010. — PMID 21381061.
- ↑ Stoltzfus A. On the possibility of constructive neutral evolution. (англ.) // Journal Of Molecular Evolution. — 1999. — August (vol. 49, no. 2). — P. 169—181. — PMID 10441669.
Эта статья входит в число хороших статей русскоязычного раздела Википедии. |